南湖新闻网讯(通讯员 胡昊)近日,加州大学河滨分校陈雪梅课题组与我校园艺林学学院联合培养博士后胡昊在植物RNA 5’端修饰研究上取得新进展,该研究报道了一种可广泛应用于原核与真核生物的RNA 5’端NAD+修饰转录本的检测技术。
RNA化学修饰是目前表观遗传学研究的热点领域,迄今报道的RNA修饰超过100种,并且广泛分布在mRNA以及其他非编码RNA上,对RNA的生物功能有着重要的影响。生物mRNA中最重要的一种修饰为5’端帽子修饰,在真核生物中主要为7-甲基鸟苷酸(m7G)结构。这个结构行使许多重要的RNA分子生物学过程,包括多聚腺苷酸化,mRNA前体剪切,mRNA出核运输,维持转录本稳定以及启动翻译等等,最终影响到基因以及蛋白的时空分布。
长期以来,m7G是真核生物唯一的帽子修饰,但是近几年研究发现,生物体内参与氧化还原反应的关键代谢中间物质烟酰胺腺嘌呤二磷酸NAD+同样也可作为RNA 5’端帽子修饰。从2015年在细菌中首次鉴定到相应的NAD+修饰开始,已经陆续在酵母,哺乳动物,人类细胞,拟南芥中检测到NAD-RNA转录本。而目前常用的NAD-RNA的检测方法主要通过酶催化化学反应对NAD-RNA进行富集并结合高通量测序的NAD captureSeq技术。然而此过程中金属离子Cu2+的参与会导致RNA的严重降解从而大大降低检测效率以及导致RNA全长信息丢失,并且该反应对于m7G-RNA同样具备非特异性富集从而影响NAD-RNA的检测准确性。
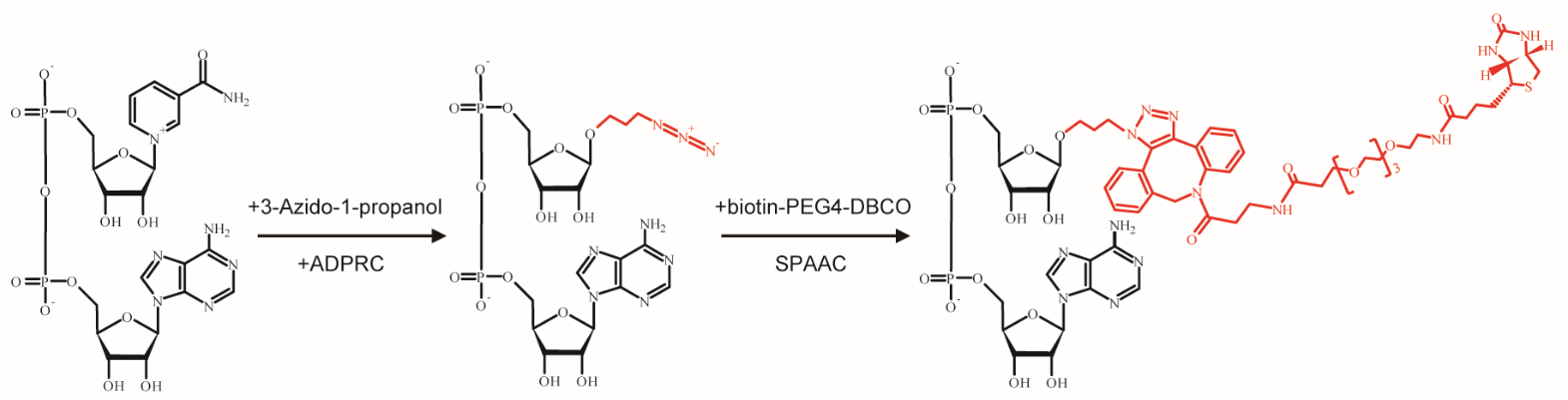
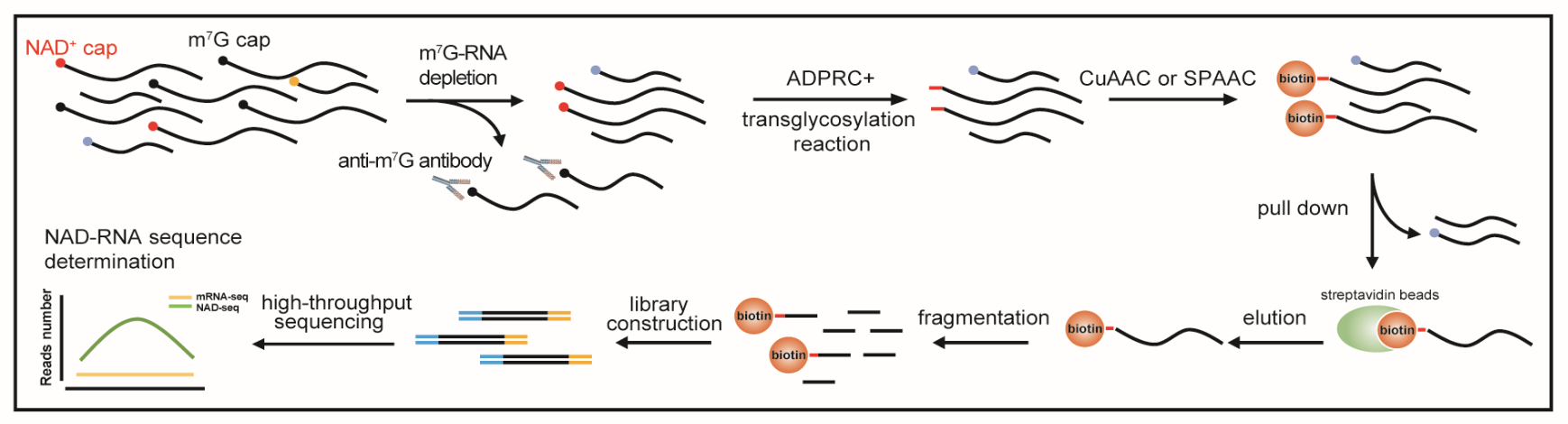
该研究开发了一种无Cu2+催化的SPAAC-NAD-seq检测技术,在通过免疫沉淀手段去除m7G-RNA的基础上,利用二磷酸腺苷核糖基环化酶(ADPRC)催化反应结合叠氮化物-炔烃环加成反应(SPAAC)富集NAD-RNA,并进一步进行高通量测序。该SPAAC-NAD-seq技术较传统的NAD captureSeq技术更为灵敏和准确,并且能保留RNA的全长信息。通过此技术,研究人员在拟南芥中检测到了5642个NAD-RNA转录本,其中包括两千多个之前并未鉴定到的低表达水平的NAD-RNA,这些转录本主要来源于核基因组和线粒体基因组,并被预测主要参与部分生物与非生物胁迫响应、氧化还原反应等生物学过程。
该研究为在基因组水平上鉴定原核生物与真核生物体内NAD-RNA提供了准确高效的检测手段,为进一步揭示此新型NAD帽子修饰在RNA上的分子功能及其潜在的生物学意义提供了技术支持和理论创新。
审核人:产祝龙
【英文摘要】
Nicotinamide adenine diphosphate (NAD+) is a novel messenger RNA 5′ cap in Escherichia coli, yeast, mammals, and Arabidopsis. Transcriptome-wide identification of NAD+-capped RNAs (NAD-RNAs) was accomplished through NAD captureSeq, which combines chemoenzymatic RNA enrichment with high-throughput sequencing. NAD-RNAs are enzymatically converted to alkyne-RNAs that are then biotinylated using a copper-catalyzed azide–alkyne cycloaddition (CuAAC) reaction. Originally applied to E. coli RNA, which lacks the m7G cap, NAD captureSeq was then applied to eukaryotes without extensive verification of its specificity for NAD-RNAs vs. m7G-capped RNAs (m7G-RNAs). In addition, the Cu2+ ion in the CuAAC reaction causes RNA fragmentation, leading to greatly reduced yield and loss of full-length sequence information. We developed an NAD-RNA capture scheme utilizing the copper-free, strain-promoted azide–alkyne cycloaddition reaction (SPAAC). We examined the specificity of CuAAC and SPAAC reactions toward NAD-RNAs and m7G-RNAs and found that both prefer the former, but also act on the latter. We demonstrated that SPAAC-NAD sequencing (SPAAC-NAD-seq), when combined with immunodepletion of m7G-RNAs, enables NAD-RNA identification with accuracy and sensitivity, leading to the discovery of new NAD-RNA profiles in Arabidopsis. Furthermore, SPAAC-NAD-seq retained full-length sequence information. Therefore, SPAAC-NAD-seq would enable specific and efficient discovery of NAD-RNAs in prokaryotes and, when combined with m7G-RNA depletion, in eukaryotes.
